Single Cell RNA Sequencing(PanoCell)
單細胞測序技術(Single Cell Sequencing)是在單個細胞水平上,揭示全基因組范圍內的所有基因表達情況,包括鑒定組織細胞類型,反映不同樣本間的細胞異質性和組織微環境。
【烈冰智造】于2021年9月發布了第一款用于單細胞測序的實驗耗材——PanoCell®雙通道微孔芯片;2022年9月推出了靈活度更高、兼容性更好的PanoCell®微孔芯片V2.0版。升級后的V2.0芯片將提供自研蜂巢板+BD平臺原裝進口單細胞分選試劑+華大T7超高通量測序儀的服務新方案!

1. 自主研發PanoCell微孔芯片,搭配BD Rhapsody單細胞平臺+BD磁珠+BD全套原裝試劑及protocol;
4.完善的質控流程和數據分析經驗:烈冰自研的CytoNavigator 百萬級通量數據分析系統確保數據的可靠性和質量,同時能夠提供個性化、定制化的數據分析服務,幫助研究人員從海量的單細胞數據中獲取有意義的信息。
樣本類型:
組織、血液、培養的細胞系、制備好的單細胞懸液
注:若客戶樣本為組織,且無能力進行組織解離來獲取單細胞懸液,烈冰將盡可能提供技術及實驗上的幫助,但因不同類型樣本的特異性,無法保證實驗方法適用于所有類型組織。
質量要求:
1. 細胞活性大于70%
2. 濃度為500-2000細胞/μl
3. 體積不小于200μl
4. 細胞培養基及緩沖液不能含Ca2+和Mg2+
5. 細胞體積小于40μm






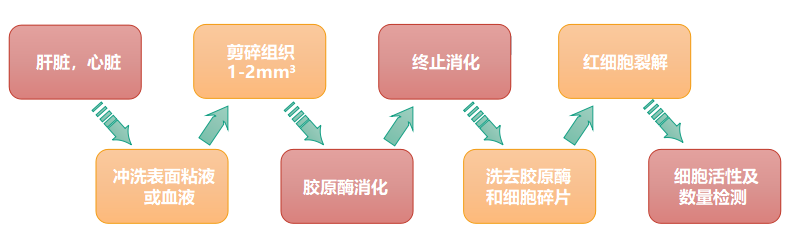
1. 單細胞懸液制備:根據樣本特性選擇合適的單細胞懸液制備方法,注意紅細胞裂解;如若客戶樣本為已經制備好單細胞懸液,該步驟可以省略;
2. 細胞活性檢測:細胞活性需大于70%;
3. 單細胞捕獲:通過分選平臺(BD)對每個細胞進行捕獲;
4. 細胞/轉錄本標簽添加:對結合磁珠標簽的RNA進行逆轉錄引入CB和UMI;
5. 文庫構建:對cDNA進行隨機引物PCR擴增;
6. 上機測序:烈冰推薦Illumina Hiseq或NovaSeq測序平臺,數據量100G/樣本。


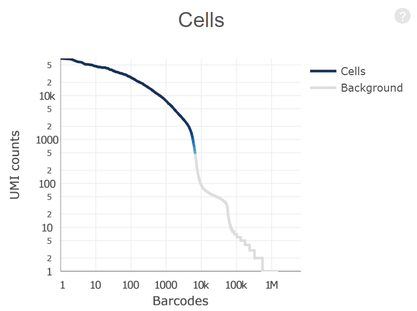
BD Rhapsody?單細胞分析系統的誕生基于 BD 在細胞生物學領域 40年的專業技術, 采用CytoSeq技術進行單細胞捕獲。應用烈冰PanoCell微孔芯片(包含20W+的微孔(該數量級遠大于Input細胞數量)),搭配BD平臺磁珠+全套試劑+protocol,保證單孔中的單細胞捕獲。同時避免了傳統微流控系統中存在的概率碰撞影響捕獲效率的問題,采用微孔捕獲相對會有更好的捕獲效率,保證Input細胞的全面使用。

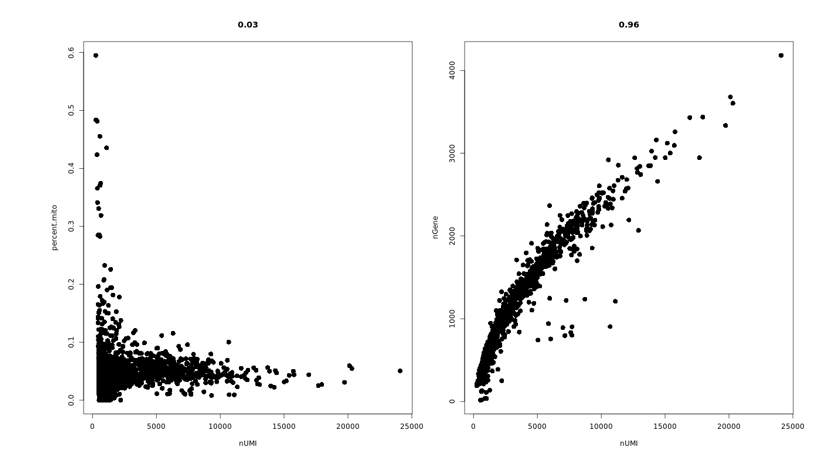
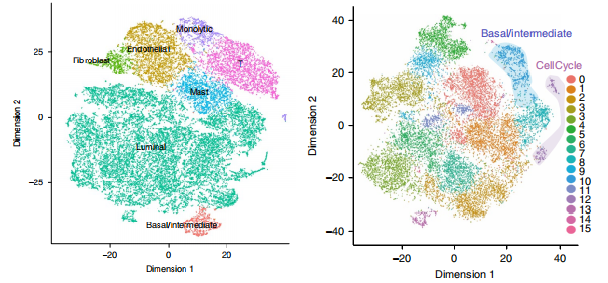
Chen, et al.Nature Cell Biology, 2021 Jan;23(1):87-98.
注:左圖為前列腺腫瘤組織細胞亞群鑒定t-SNE圖展示;右圖細胞亞群分群情況
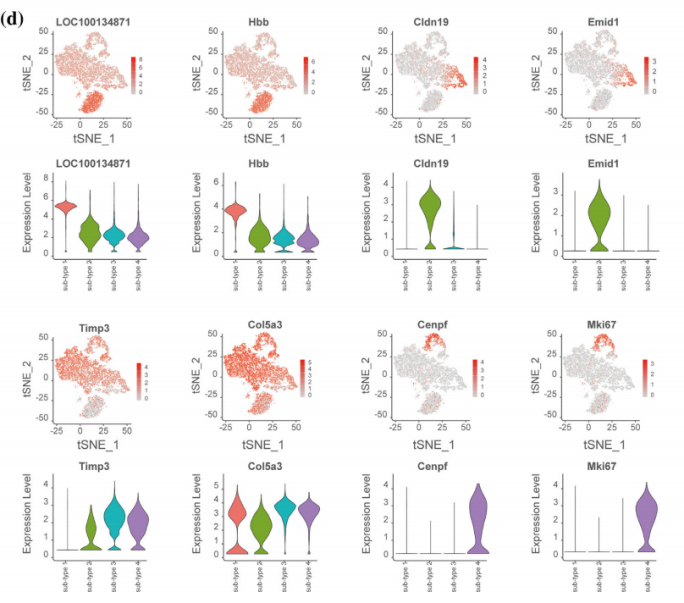
Zhang et al., Glia. 2021 Mar;69(3):765-778.
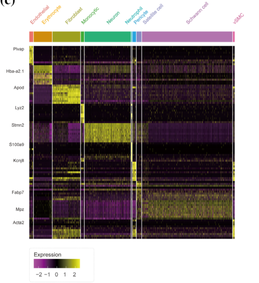
Zhang, et al. Glia. 2021 Mar;69(3):765-778.
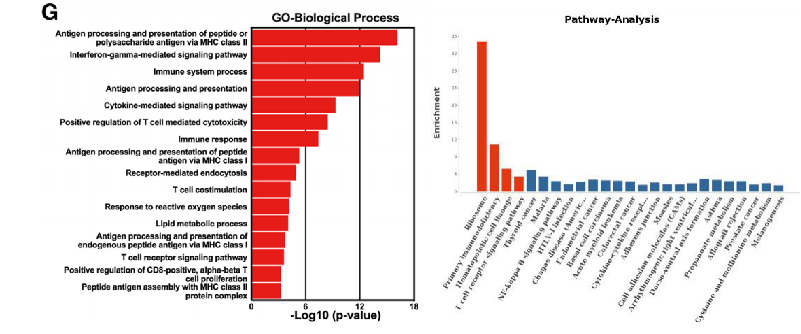
Zhang C, et al. J Immunother Cancer 2021;9:e002312.
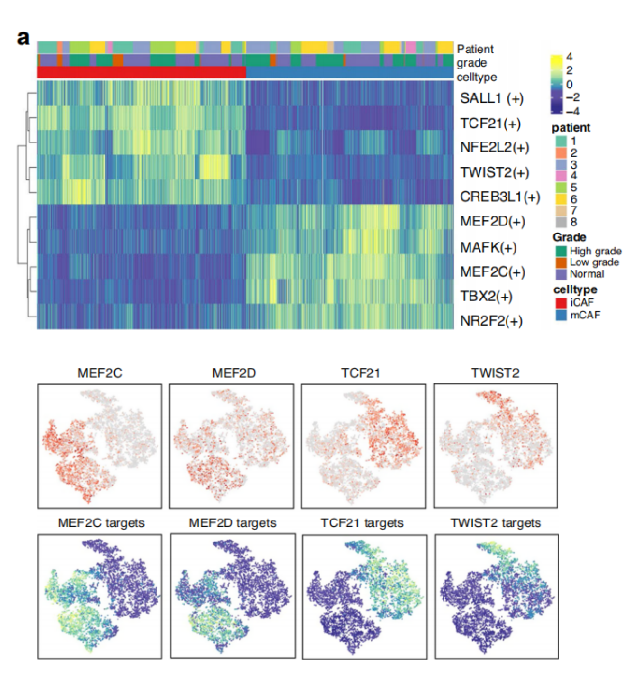
Chen, et al. Nature Communication 2020; 11:5077
注:該圖為不同cluster之間轉錄因子調控強度heatmap圖
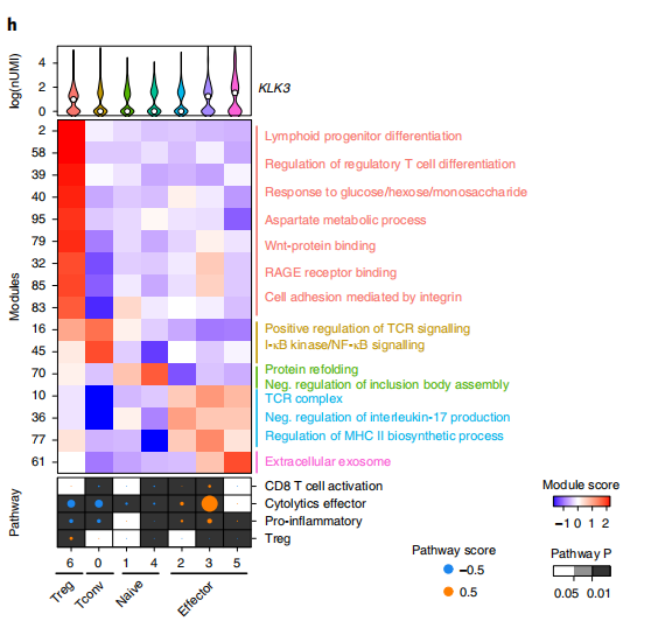
Chen, et al.Nature Cell Biology, 2021 Jan;23(1):87-98.
注:該圖為不同cluster之間信號同理調控強度熱圖
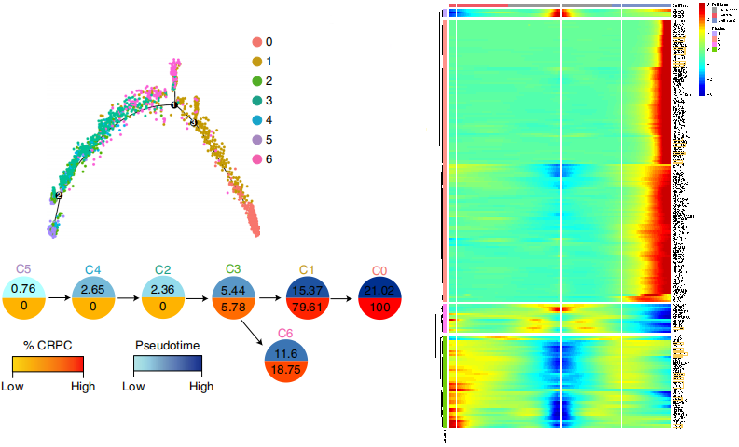
Chen, et al.Nature Cell Biology, 2021 Jan;23(1):87-98.
注:細胞間狀態轉換的pesudotime軌跡圖(左)、軌跡中細胞占比餅圖 和 Heatmap圖(右)

Chen, et al. Nature Communication 2020; 11:5077
注:左圖:橫坐標表示細胞類型,縱坐標表示細胞間通訊配受體關系,圓圈大小表示顯著性差異水平,圓圈顏色越紅表示細胞間通訊關系越強
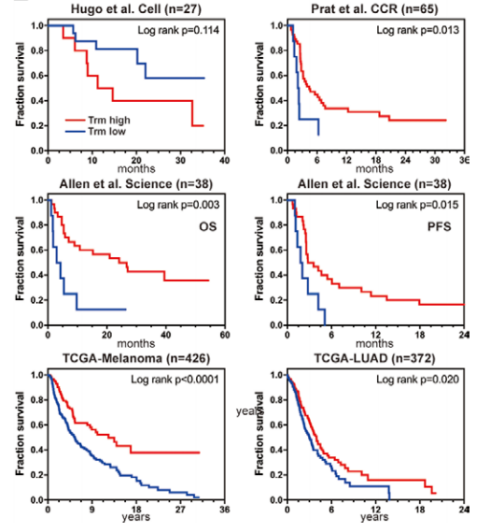
Zhang C, et al. J Immunother Cancer 2021;9:e002312.
[1] CD_99 G1 neutrophils modulate osteogenic differentiation of mesenchymal stem cells in the pathological process of ankylosing spondylitis.Ann Rheum Dis.2023 Oct;IF=27.4
[2] Immunosuppressive CD10+ALPL+ neutrophils promote resistance to anti-PD-1therapy in HCC by mediating irreversible exhaustion of T cells. Journal of Hepatology. 2023 Aug;IF=25.7
[3] Endothelial response to type I interferon contributes to vasculopathy and fibrosis and predicts disease progression of systemic sclerosis.Arthritis & Rheumatology. 2023 Jul; IF=13.3
[4] Schwann cells regulate tumor cells and cancer-associated fibroblasts in the pancreatic ductal adenocarcinoma microenvironment. Nature Communicationsl. 2023 Jul ; IF=16.6
[5] WT1+ glomerular parietal epithelial progenitors promote renal proximal tubule regeneration after severe acute kidney injury. 2023 Feb; IF=12.4
[6] Single-cell RNA-seq analysis reveals BHLHE40-driven pro-tumour neutrophils with hyperactivated glycolysis in pancreatic tumour microenvironment. GUT. 2022 Jun; IF=24.5