Whole Transcriptome Sequencing
全轉(zhuǎn)錄組是指特定細(xì)胞在特定狀態(tài)下所能轉(zhuǎn)錄出來的所有RNA的總和,包括mRNA和非編碼RNA(non-coding RNA)。針對(duì)非編碼RNA的研究主要集中在具有調(diào)控作用的miRNA,lncRNA和circRNA。基于二代測(cè)序技術(shù)的全轉(zhuǎn)錄組測(cè)序研究,同時(shí)分析同一樣本中的mRNA,lncRNA,circRNA,miRNA,并且通過兩兩關(guān)聯(lián)分析、三元關(guān)聯(lián)分析、多元關(guān)聯(lián)分析,使研究內(nèi)容更加系統(tǒng)化,致力于深入挖掘生命現(xiàn)象背后的轉(zhuǎn)錄調(diào)控問題。

ceRNA(競(jìng)爭(zhēng)性內(nèi)源RNA)機(jī)制示意圖
Wang Y et al., Trends Genet, 2016
1. 雙文庫構(gòu)建:small RNA文庫和去核糖體的鏈特異性文庫,烈冰 8年建庫經(jīng)驗(yàn),保證建庫質(zhì)量;
2. 4種RNA全方位分析:不僅能定量分析已知的lncRNA和miRNA,還能通過Stringtie重建轉(zhuǎn)錄本預(yù)測(cè)新的lncRNA;并通過預(yù)測(cè)的circRNA進(jìn)行靶向分析 ,從而得到miRNA-mRNA、lncRNA-miRNA、以及circRNA-miRNA的靶向關(guān)系;
3. 數(shù)據(jù)庫全面整合:整合并定時(shí)升級(jí)生物學(xué)領(lǐng)域內(nèi)公認(rèn)數(shù)據(jù)庫和靶基因預(yù)測(cè)算法,如NP Inter、miRBase、RNAhybrid等,保證分析結(jié)果緊跟行業(yè)前沿;
4. 上游測(cè)序+下游驗(yàn)證:客戶只需提供細(xì)胞,組織或者總RNA,烈冰為您完成從上機(jī)測(cè)序到數(shù)據(jù)分析整套服務(wù)流程,同時(shí)可進(jìn)行后續(xù)qPCR驗(yàn)證。
組織樣品:
1. 動(dòng)物組織≥1g;
2. 植物組織≥2g;
3. 細(xì)胞樣品≥1×106個(gè);
4. 全血≥5mL;
5. 菌體≥106個(gè)或≥30mg。
RNA樣品:
1. 樣品需求量: RNA≥10 μg;
2. 樣品濃度:RNA樣品≥100 ng/μl;
3. 樣品純度:OD260/OD280在1.8-2.2之間,OD260/OD230≥2,28S/18S≥1,動(dòng)物樣品RIN≥7.0,植物樣品RIN≥6.5,RNA無明顯降解。





1. 客戶樣本:細(xì)胞量在106以上;
2. 總RNA提取及質(zhì)控:凝膠電泳質(zhì)控→Nanodrop質(zhì)控→Agilent 2200質(zhì)控;
3. small RNA文庫構(gòu)建:切膠范圍10-50bp,單端測(cè)序SE50,文庫分子18-30bp;
4. 去核糖體文庫構(gòu)建:逆轉(zhuǎn)錄后用RNase處理,去除rRNA;
5. 上機(jī)測(cè)序:烈冰建議選擇NovaSeq,雙端測(cè)序,通量大,堿基精度高,而且成本低,速度快。



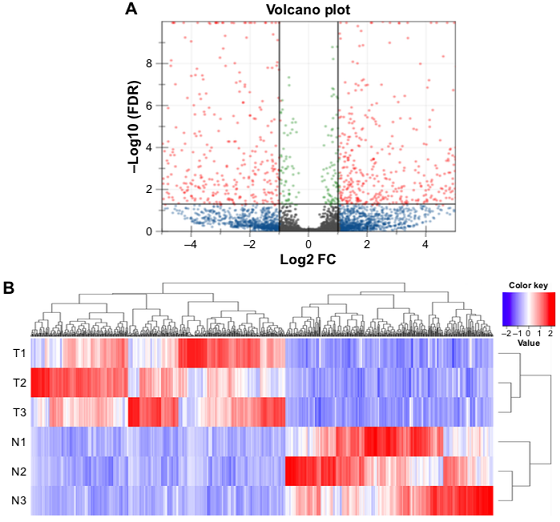
差異lncRNAs火山圖分析和聚類分析
Yang F et al., Gene, 2016
注:(A)差異lncRNA火山圖分析結(jié)果,紅色表示顯著差異的lncRNA,藍(lán)色表示非顯著差異的lncRNA;(B)差異lncRNA聚類分析的Heat map,紅色越深表示lncRNA上調(diào)越顯著,藍(lán)色越深表示lncRNA下調(diào)越顯著。
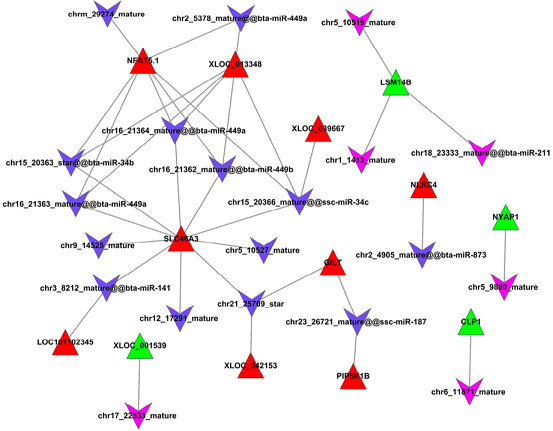
lncRNA-miRNA靶向作用關(guān)系
Miao X et al., Sci Rep, 2016
注:紅色三角表示上調(diào)lncRNAs,綠色三角表示下調(diào)lncRNAs,紫色V型三角表示上調(diào)miRNAs,藍(lán)色V型三角表示下調(diào)miRNAs。
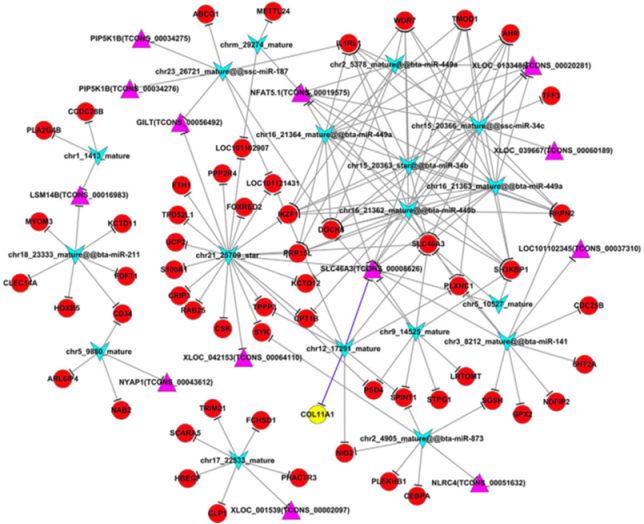
lncRNA-miRNA-mRNA Network
Miao X et al., Sci Rep, 2016
注:紫色三角形表示lncRNA,紅色圓點(diǎn)表示mRNA,黃色圓點(diǎn)表示關(guān)鍵mRNA,藍(lán)色V型三角表示miRNA。
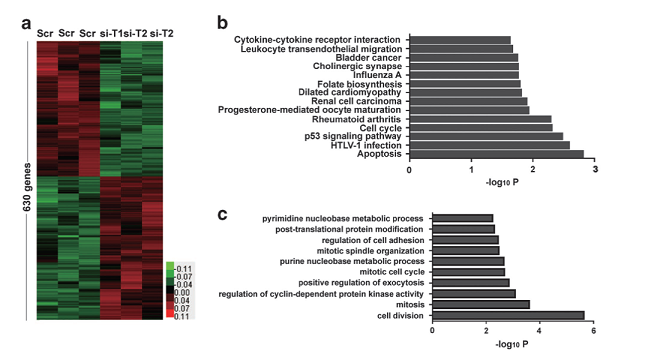
差異基因GO分析和Pathway分析
Xu T et al., Oncogene. 2015
注:(a)lncRNA TINCR-siRNA VS scrambled siRNA差異基因聚類分析圖;(b)差異基因顯著富集的pathway條目;(c)差異基因顯著富集的GO條目。
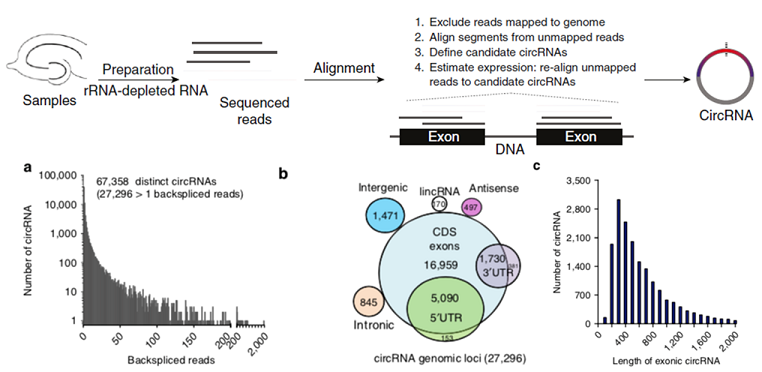
circRNA預(yù)測(cè)工作流和預(yù)測(cè)結(jié)果(正常組織 VS 癌組織)
Chen W et al., Nat Neurosci. 2015/Zheng Q et al., Nature Communications, 2016
注:(a)橫坐標(biāo)表示circRNA反向剪接reads數(shù),縱坐標(biāo)表示circRNA數(shù)量;(b)circRNA在基因組結(jié)構(gòu)上的分布;(c)橫坐標(biāo)表示exonic circRNA長度,縱坐標(biāo)表示circRNA數(shù)量。
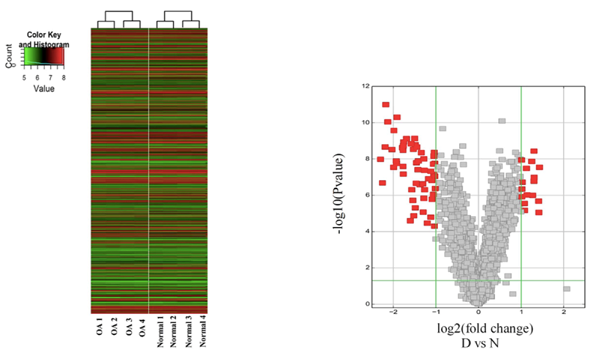
差異circRNA分析結(jié)果
Liu Q et al., Scientific Reports, 2016
注:左圖為差異circRNA的聚類分析圖(OA VS normal軟骨組織);右圖為差異circRNA的火山圖,紅點(diǎn)表示差異顯著的circRNA。
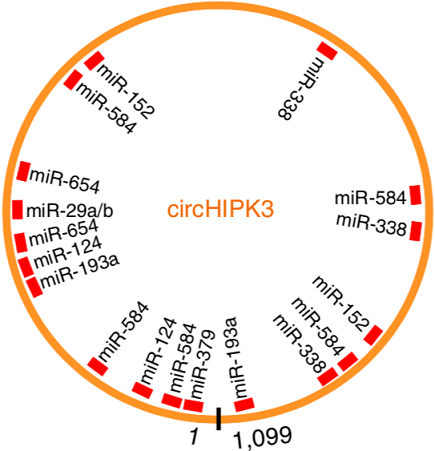
circRNA-miRNA相互作用
Zheng Q et al., Nature Communications, 2016
注:該圖展示了circHIPK3相互作用的miRNAs的假定結(jié)合位點(diǎn)。
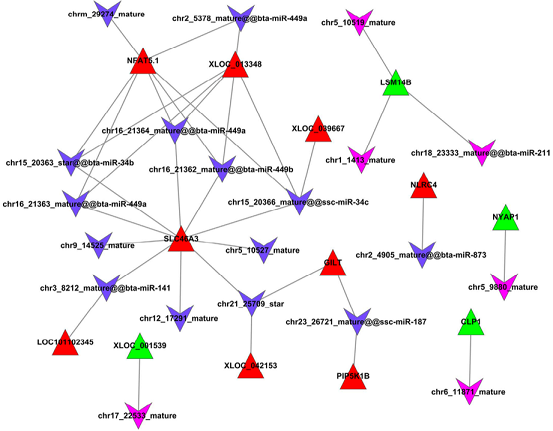
circRNA-miRNA-mRNA Network
Liu Q et al., Scientific Reports, 2016
注:綠色圓點(diǎn)表示circRNA,黃色菱形表示mRNA,紫色V型三角表示miRNA。
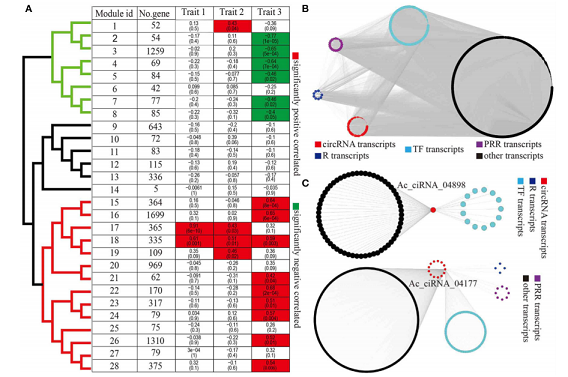
circRNA和蛋白編碼基因的WGCNA分析
Wang Z et al. Frontiers in plant science, 2017
注:該圖為WGCNA計(jì)算出來的不同module,與不同表型相關(guān)性高的module,并對(duì)moudle16以及兩個(gè)circRNA的關(guān)系網(wǎng)絡(luò)圖分別進(jìn)行了可視化展示。
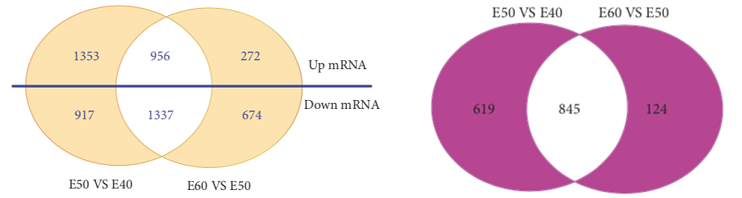
E50 VS E40和E60 VS E50差異mRNA/lncRNA韋恩圖
Li Y et al., BioMed Research International. 2019
注:左圖為E50 VS E40差異mRNA和E60 VS E50差異mRNA的韋恩分析結(jié)果;右圖為E50 VS E40差異lncRNA和E60 VS E50差異lncRNA的韋恩分析結(jié)果。
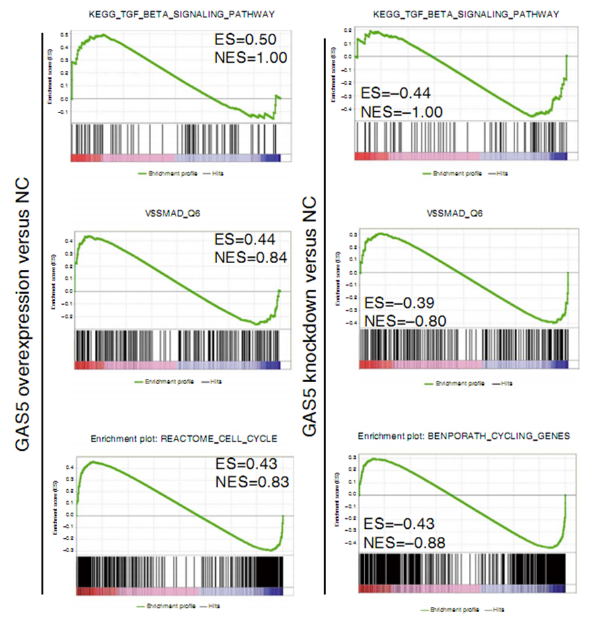
干預(yù)LncRNA GAS5的基因富集分析
Liu et al., Nat Commun, 2016
注:該圖為LncRNA GAS5過表達(dá)和敲低后,hESCs的基因富集分析結(jié)果。其中,ES表示富集度得分,NES表示ES標(biāo)化后的值,得分越高表示該基因類別與該干預(yù)呈正相關(guān)。
[1] Lei B, Zhou J, Xuan X, et al. Circular RNA expression profiles of peripheral blood mononuclear cells in hepatocellular carcinoma patients by sequence analysis. Cancer Med. 2019 Feb 4. (IF=3.202)
[2] Li Y, Li GQ, Wang F, et al. Integrated Analysis of LncRNA-mRNA Coexpression in the Extracellular Matrix of Developing Deciduous Teeth in Miniature Pigs. BioMed Research International. 2019 Jan 23. (IF=2.583)
[3] Yu Y, Zhang M, Liu J, et al. Long Non-coding RNA PVT1 Promotes Cell Proliferation and Migration by Silencing ANGPTL4 Expression in Cholangiocarcinoma. Mol Ther Nucleic Acids. 2018 Dec 7;13:503-513. (IF=5.66)
[4] Qu S, Hao X, Song W, et al. Circular RNA circRHOT1 is upregulated and promotes cell proliferation and invasion in pancreatic cancer. Epigenomics. 2018 Nov 16;11(1):53-63. (IF=4.979)
[5] Yu Y, Zhang M, Wang N, et al. Epigenetic silencing of tumor suppressor gene CDKN1A by oncogenic long non-coding RNA SNHG1 in cholangiocarcinoma. Cell Death Dis. 2018 Jul 3; 9(7):746-758. (IF=5.638)
[6] Yin D, Lu X, Su J, et al. Long noncoding RNA AFAP1-AS1 predicts a poor prognosis and regulates non-small cell lung cancer cell proliferation by epigenetically repressing p21 expression. Mol Cancer. 2018 May 24;17(1):92. (IF=7.776)
[7] Liang Ding,et al. A novel stromal lncRNA signature reprograms fibroblasts to promote the growth of oral squamous cell carcinoma via LncRNA-CAF/interleukin-33. Carcinogenesis. 2018 Mar 8;39(3):397-406. (IF=5.105)
[8] Sun D,et al.LncRNA GAS5 inhibits microglial M2 polarization and exacerbates demyelination. EMBO Rep. 2017 Oct;18(10):1801-1816. (IF=8.568)
[9] Lai, Z.Y. et al. Analysis of co-expression networks for circular RNAs and mRNAs reveals that circular RNAs hsa_circ_0047905, hsa_circ_0138960 and hascircRNA7690-15 are candidate oncogenes in gastric cancer. Cell Cycle. 2017 Oct 5:1-11. (IF=3.53)
[10] Zhang E, et al. H3K27 acetylation activated-long non-coding RNA CCAT1 affects cell proliferation and migration by regulating SPRY4 and HOXB13 expression in esophageal squamous cell carcinoma. Nucleic Acids Res. 2017 Apr 7;45(6):3086-3101.. (IF=10.162)
[11] Xu C, et al. Long non-coding rna gas5 control human embryonic stem cell self renewal by maintaining nodal signaling. Nat Commun. 2016 Nov 4;7:13287. (IF=12.124)
[12] Zheng Q, et al. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs. Nat Commun. 2016 Apr 6;7:11215. (IF=11.47)
[13] Liu Q, et al. Circular RNA Related to the Chondrocyte ECM Regulates MMP13 Expression by Functioning as a MiR-136 ‘Sponge’ in Human Cartilage Degradation. Sci Rep. 2016 Mar 2;6:22572. (IF=5.578)