Single Cell Sequencing
單細胞測序技術(single cell sequencing)是指在單個細胞水平上,對基因組、轉錄組、表觀組進行高通量測序分析的一項新技術,它能夠彌補傳統高通量測序的局限性,揭示單個細胞的基因結構和基因表達狀態,反映細胞間的異質性。
2013年,《Science》將其列為年度最值得關注的六大領域榜首;2015年再次登上Science轉化醫學封面。2018年躋身Science 年度突破技術; 2019年,單細胞多組學被列為Nature Methods年度技術,2021年,又被評選為10大創新生物科技之一...目前,單細胞測序技術在腫瘤、發育生物學、微生物學、神經科學、以及植物學等領域發揮重要作用,正成為生命科學研究的焦點,具有廣闊的應用前景。


單細胞轉錄組測序流程--點我了解更多
1. 單細胞測序技術:公司擁有行業認可的10X Genomics、BD Rhapsody以及烈冰自研的單細胞平臺,實現高質高效的單細胞測序。
2.多樣化的樣本處理經驗:公司具有豐富的單細胞懸液制備經驗,50+組織類型,100+細胞類型、10000+樣本處理經驗,確保細胞的活性能夠達到測序要求,從而獲得可靠的單細胞數據。
(圖片來源于網絡)
3.高效的文庫構建技術:公司擁有成熟的單細胞測序文庫構建技術和自動化建庫平臺,一次性完成1000-10000個細胞的文庫構建。
4.完善的質控流程和數據分析經驗:烈冰自研的CytoNavigator 百萬級通量數據分析系統確保數據的可靠性和質量,同時能夠提供個性化、定制化的數據分析服務,幫助研究人員從海量的單細胞數據中獲取有意義的信息。
樣本類型:
組織、血液、培養的細胞系、制備好的單細胞懸液
注:若客戶樣本為組織,且無能力進行組織解離來獲取單細胞懸液,烈冰將盡可能提供技術及實驗上的幫助,但因不同類型樣本的特異性,無法保證實驗方法適用于所有類型組織。
質量要求:
1. 細胞活性大于70%
2. 濃度為500-2000細胞/μl
3. 體積不小于200μl
4. 細胞培養基及緩沖液不能含Ca2+和Mg2+
5. 細胞體積小于40μm






1. 單細胞懸液制備:根據樣本特性選擇合適的單細胞懸液制備方法,注意紅細胞裂解;如若客戶樣本為已經制備好單細胞懸液,該步驟可以省略;
2. 細胞活性檢測:細胞活性需大于70%;
3. 單細胞捕獲:通過分選平臺(BD、10X、Drop-seq)對每個細胞進行捕獲;
4. 細胞/轉錄本標簽添加:對結合磁珠標簽的RNA進行逆轉錄引入CB和UMI;
5. 文庫構建:對cDNA進行隨機引物PCR擴增;
6. 上機測序:烈冰推薦Illumina Hiseq或NovaSeq測序平臺,數據量100G/樣本。

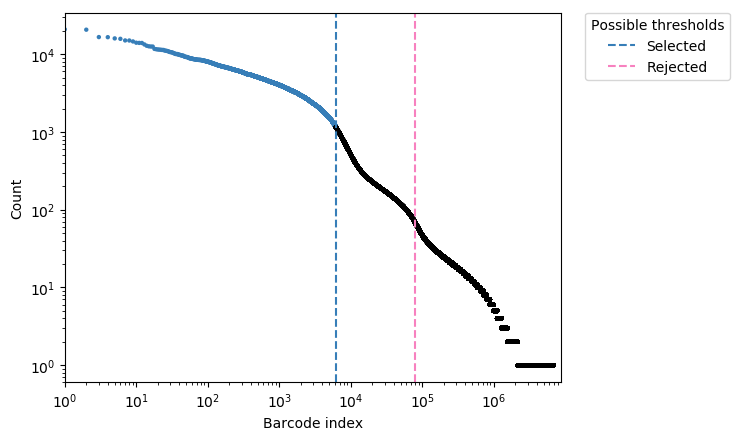
注:橫坐標為細胞數量,縱坐標為每個細胞對應的平均counts數,根據曲線的斜率判斷實際檢測的細胞數量
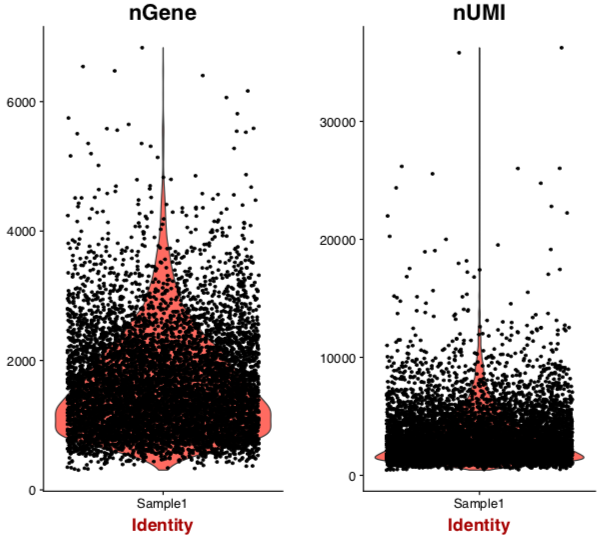
注:左圖為細胞中總共檢測到的基因數量,右圖為去除重復UMI后統計的基因數量
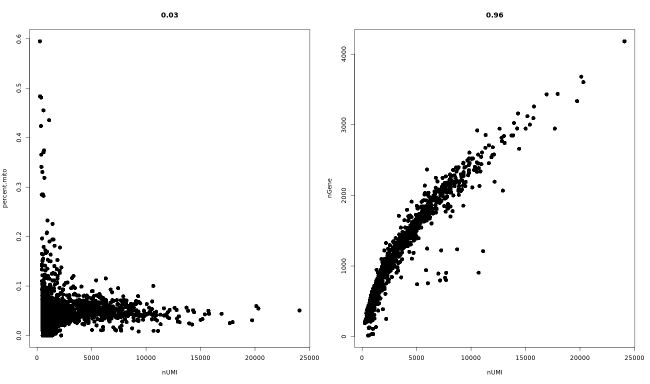
注:左圖為每個細胞中的線粒體占比——nUMI數量相關性分析圖,
右圖為單細胞測序中每一個細胞的基因——nUMI數量相關性分析圖
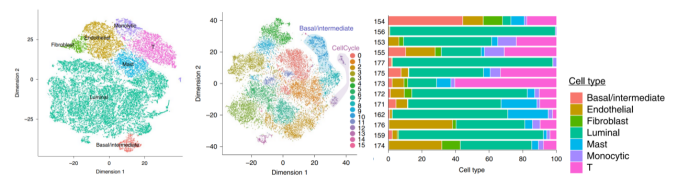
Chen, et al. Nature Cell Biology, 2021 Jan;23(1):87-98.
注:左圖為前列腺腫瘤組織細胞亞群鑒定t-SNE圖展示;右圖為不同樣本中各細胞亞群的占比情況
Zhang, et al. Glia, 2021 Mar;69(3):765-778.
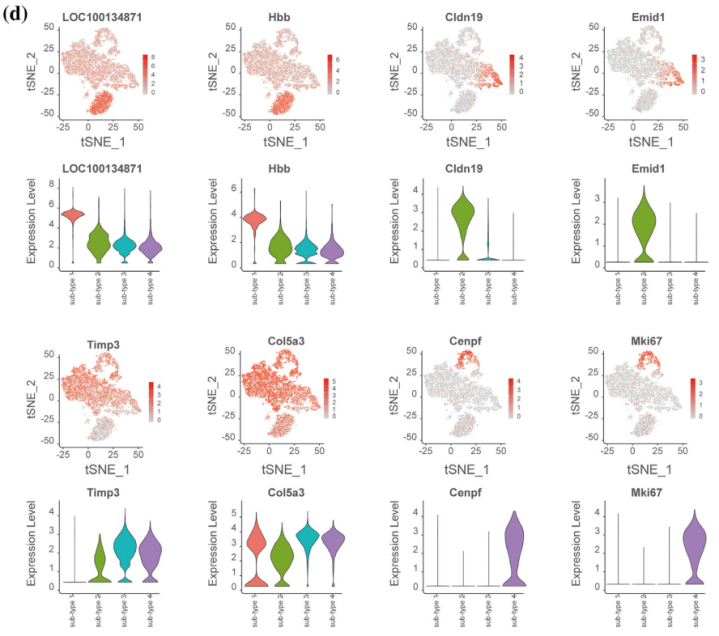
注: Marker基因的Feature Plot圖和Violin圖
Zhang, et al. Glia, 2021 Mar;69(3):765-778.
Zhang C, et al. J Immunother Cancer, 2021;9:e002312.
Chen, et al. Nature Cell Biology, 2021 Jan;23(1):87-98.
Chen, et al. Nature Communication 2020; 11:5077
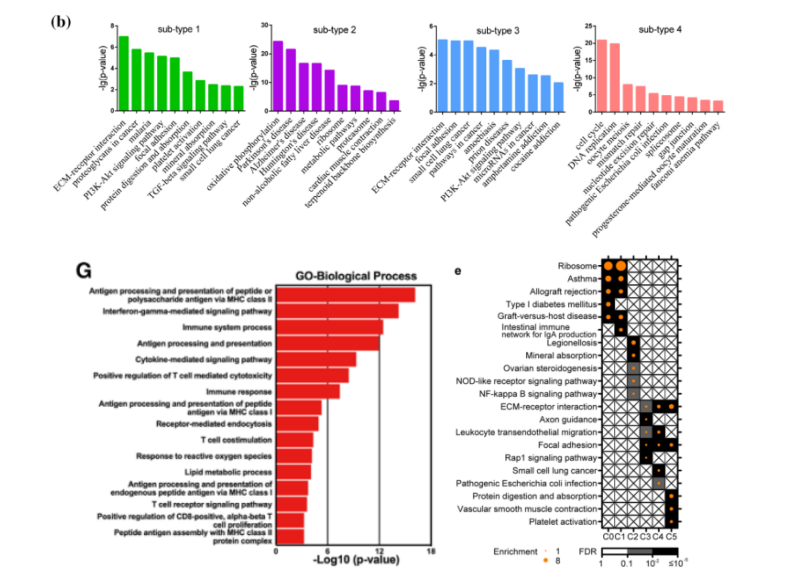
Chen, et al. Nature Cell Biology, 2021 Jan;23(1):87-98.
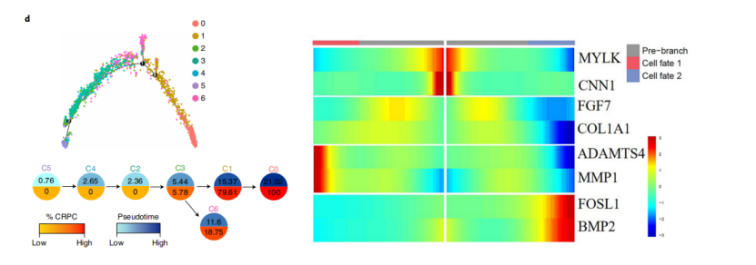
Chen, et al. Nature Cell Biology, 2021 Jan;23(1):87-98.
Sun, et al. Annals of the Rheumatic Diseases, 2019, 79(3):215926.
注:細胞間狀態轉換的pesudotime軌跡圖(左)和Heatmap圖(右)
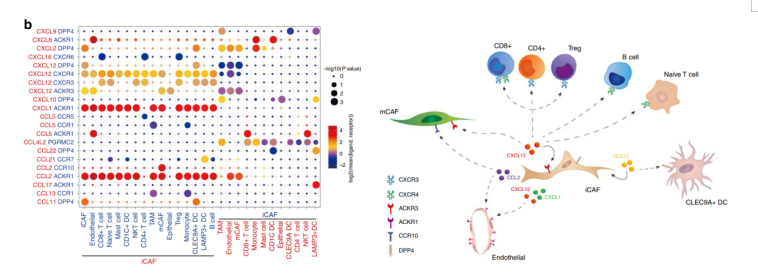
Chen, et al. Nature Communication, 2020;11:5077.
注:橫坐標表示細胞類型,縱坐標表示細胞間通訊關系,圓圈大小表示顯著性差異水平,圓圈顏色越紅表示細胞間通訊關系越強
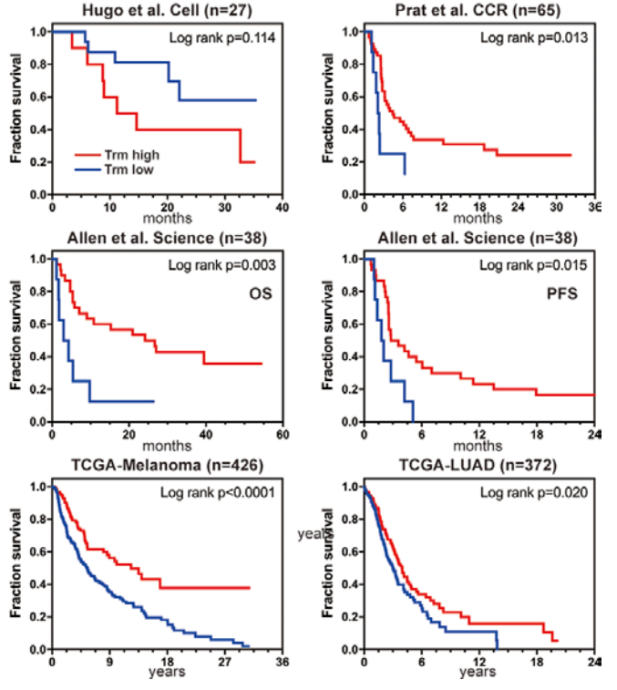
Zhang C, et al. J Immunother Cancer, 2021;9:e002312.
注:橫坐標表示生存期,縱坐標表示占比,不同顏色曲線代表不同分組
烈冰助力文獻:
[1] Antiandrogen treatment induces stromal cell reprogramming to promote castration resistance in prostate cancer,Cancer cell,2023 IF=50.3
[2] A multimorphic mutation in IRF4 causes human autosomal dominant combined immunodeficiency,Science Immunology,2023 IF=24.8
[3] Tumor-intrinsic YTHDF1 drives immune evasion and resistance to immune checkpoint inhibitors via promoting MHC-I degradation,Nature Communications,2023,IF=16.6
[4] ECSIT Is a Critical Factor for Controlling Intestinal Homeostasis and Tumorigenesis through Regulating the Translation of YAP Protein,Advanced Science,2023,IF=15.1
[5] Systemic immune dysregulation in severe tuberculosis patients revealed by a single-cell transcriptome atlas,Journal of Infection,2023,IF=28.2
[6] Single-cell and spatial dissection of precancerous lesions underlying the initiation process of oral squamous cell carcinoma,Cell Discovery,2023,IF=33.5
[7] Immunosuppressive CD10+ALPL+ neutrophils promote resistance to anti-PD-1therapy in HCC by mediating irreversible exhaustion of T cells, Journal of Hepatology,2023,IF=25.7
[8] CD_99 G1 neutrophils modulate osteogenic differentiation of mesenchymal stem cells in the pathological process of ankylosing spondylitis,Annals of the Rheumatic Diseases,2023 IF=27.4
[9] Targeting VEGF-A/VEGFR2 Y949 Signaling Mediated Vascular Permeability Alleviates Hypoxic Pulmonary Hypertension,Circulation,2022 IF=37.8
[10] A rice single cell transcriptomic atlas defines the developmental trajectories of rice floret and inflorescence meristems,New Phytologist,2022 IF=10.15